PyCaret是一个开源、低代码Python机器学习库,能够自动化机器学习工作流程。它是一个端到端的机器学习和模型管理工具,极大地加快了实验周期,提高了工作效率。PyCaret本质上是围绕几个机器学习库和框架(如scikit-learn、XGBoost、LightGBM、CatBoost、spaCy、Optuna、Hyperopt、Ray等)的Python包装器,与其他开源机器学习库相比,PyCaret可以用少量代码取代数百行代码。PyCaret开源仓库地址: pycaret ,官方文档地址为: pycaret-docs 。
PyCaret基础版安装命令如下:
pip install pycaret
完整版安装代码如下:
pip install pycaret[full]
此外以下模型可以调用GPU
- Extreme Gradient Boosting
- Catboost
- Light Gradient Boosting(需要安装 lightgbm )
- Logistic Regression, Ridge Classifier, Random Forest, K Neighbors Classifier, K Neighbors Regressor, Support Vector Machine, Linear Regression, Ridge Regression, Lasso Regression(需要安装 cuml 0.15版本以上)
# 查看pycaret版本
import pycaret
pycaret.__version__
'3.3.2'
1 快速入门
PyCaret支持多种机器学习任务,包括分类、回归、聚类、异常检测和时序预测。本节主要介绍如何利用PyCaret构建相关任务模型的基础使用方法。关于更详细的PyCaret任务模型使用,请参考:
| Topic | NotebookLink |
|---|---|
| 二分类BinaryClassification | link |
| 多分类MulticlassClassification | link |
| 回归Regression | link |
| 聚类Clustering | link |
| 异常检测AnomalyDetection | link |
| 时序预测TimeSeriesForecasting | link |
1.1 分类
PyCaret的classification模块是一个可用于二分类或多分类的模块,用于将元素分类到不同的组中。一些常见的用例包括预测客户是否违约、预测客户是否流失、以及诊断疾病(阳性或阴性)。示例代码如下所示:
数据准备
加载糖尿病示例数据集:
from pycaret.datasets import get_data
# 从本地加载数据,注意dataset是数据的文件名
data = get_data(dataset='./datasets/diabetes', verbose=False)
# 从pycaret开源仓库下载公开数据
# data = get_data('diabetes', verbose=False)
# 查看数据类型和数据维度
type(data), data.shape
(pandas.core.frame.DataFrame, (768, 9))
# 最后一列表示是否为糖尿病患者,其他列为特征列
data.head()
利用PyCaret核心函数setup,初始化建模环境并准备数据以供模型训练和评估使用:
from pycaret.classification import ClassificationExperiment
s = ClassificationExperiment()
# target目标列,session_id设置随机数种子, preprocesss是否清洗数据,train_Size训练集比例, normalize是否归一化数据, normalize_method归一化方式
s.setup(data, target = 'Class variable', session_id = 0, verbose= False, train_size = 0.7, normalize = True, normalize_method = 'minmax')
<pycaret.classification.oop.ClassificationExperiment at 0x200b939df40>
查看基于setup函数创建的变量:
s.get_config()
{'USI',
'X',
'X_test',
'X_test_transformed',
'X_train',
'X_train_transformed',
'X_transformed',
'_available_plots',
'_ml_usecase',
'data',
'dataset',
'dataset_transformed',
'exp_id',
'exp_name_log',
'fix_imbalance',
'fold_generator',
'fold_groups_param',
'fold_shuffle_param',
'gpu_n_jobs_param',
'gpu_param',
'html_param',
'idx',
'is_multiclass',
'log_plots_param',
'logging_param',
'memory',
'n_jobs_param',
'pipeline',
'Seed',
'target_param',
'test',
'test_transformed',
'train',
'train_transformed',
'variable_and_property_keys',
'variables',
'y',
'y_test',
'y_test_transformed',
'y_train',
'y_train_transformed',
'y_transformed'}
查看归一化的数据:
s.get_config('X_train_transformed')
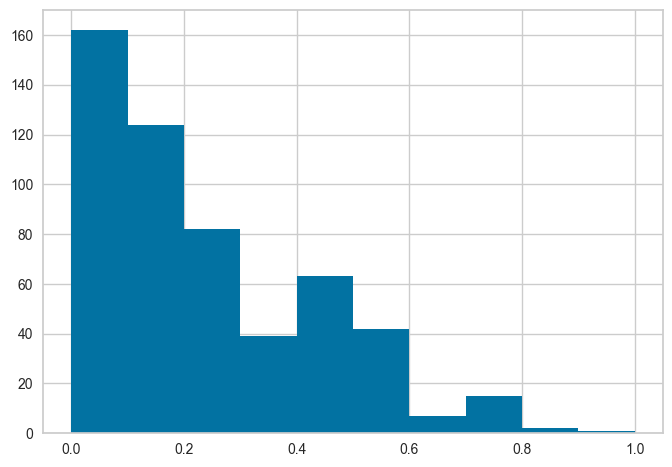
绘制某列数据的柱状图:
s.get_config('X_train_transformed')['Number of times pregnant'].hist()
<AxesSubplot:>
当然也可以利用如下代码创建任务示例来初始化环境:
from pycaret.classification import setup
# s = setup(data, target = 'Class variable', session_id = 0, preprocess = True, train_size = 0.7, verbose = False)
模型训练与评估
PyCaret提供了compare_models函数,通过使用默认的10折交叉验证来训练和评估模型库中所有可用估计器的性能:
best = s.compare_models()
# 选择某些模型进行比较
# best = s.compare_models(include = ['dt', 'rf', 'et', 'gbc', 'lightgbm'])
# 按照召回率返回n_select性能最佳的模型
# best_recall_models_top3 = s.compare_models(sort = 'Recall', n_select = 3)
| Model | Accuracy | AUC | Recall | Prec. | F1 | Kappa | MCC | TT (Sec) | |
|---|---|---|---|---|---|---|---|---|---|
| lr | Logistic Regression | 0.7633 | 0.8132 | 0.4968 | 0.7436 | 0.5939 | 0.4358 | 0.4549 | 0.2720 |
| ridge | Ridge Classifier | 0.7633 | 0.8113 | 0.5178 | 0.7285 | 0.6017 | 0.4406 | 0.4560 | 0.0090 |
| lda | Linear Discriminant Analysis | 0.7633 | 0.8110 | 0.5497 | 0.7069 | 0.6154 | 0.4489 | 0.4583 | 0.0080 |
| ada | Ada Boost Classifier | 0.7465 | 0.7768 | 0.5655 | 0.6580 | 0.6051 | 0.4208 | 0.4255 | 0.0190 |
| svm | SVM - Linear Kernel | 0.7408 | 0.8087 | 0.5921 | 0.6980 | 0.6020 | 0.4196 | 0.4480 | 0.0080 |
| nb | Naive Bayes | 0.7391 | 0.7939 | 0.5442 | 0.6515 | 0.5857 | 0.3995 | 0.4081 | 0.0080 |
| rf | Random Forest Classifier | 0.7337 | 0.8033 | 0.5406 | 0.6331 | 0.5778 | 0.3883 | 0.3929 | 0.0350 |
| et | Extra Trees Classifier | 0.7298 | 0.7899 | 0.5181 | 0.6416 | 0.5677 | 0.3761 | 0.3840 | 0.0450 |
| gbc | Gradient Boosting Classifier | 0.7281 | 0.8007 | 0.5567 | 0.6267 | 0.5858 | 0.3857 | 0.3896 | 0.0260 |
| lightgbm | Light Gradient Boosting Machine | 0.7242 | 0.7811 | 0.5827 | 0.6096 | 0.5935 | 0.3859 | 0.3876 | 0.0860 |
| qda | Quadratic Discriminant Analysis | 0.7150 | 0.7875 | 0.4962 | 0.6225 | 0.5447 | 0.3428 | 0.3524 | 0.0080 |
| knn | K Neighbors Classifier | 0.7131 | 0.7425 | 0.5287 | 0.6005 | 0.5577 | 0.3480 | 0.3528 | 0.2200 |
| dt | Decision Tree Classifier | 0.6685 | 0.6461 | 0.5722 | 0.5266 | 0.5459 | 0.2868 | 0.2889 | 0.0100 |
| dummy | Dummy Classifier | 0.6518 | 0.5000 | 0.0000 | 0.0000 | 0.0000 | 0.0000 | 0.0000 | 0.0120 |
返回当前设置中所有经过训练的模型中的最佳模型:
best_ml = s.automl()
# best_ml
# 打印效果最佳的模型
print(best)
LogisticRegression(C=1.0, class_weight=None, dual=False, fit_intercept=True,
intercept_scaling=1, l1_ratio=None, max_iter=1000,
multi_class='auto', n_jobs=None, penalty='l2',
random_state=0, solver='lbfgs', tol=0.0001, verbose=0,
warm_start=False)
数据可视化
PyCaret也提供了plot_model函数可视化模型的评估指标,plot_model函数中的plot用于设置评估指标类型。plot可用参数如下(注意并不是所有的模型都支持以下评估指标):
- pipeline: Schematic drawing of the preprocessing pipeline
- auc: Area Under the Curve
- thresHOLD: Discrimination Threshold
- pr: Precision Recall Curve
- confusion_matrix: Confusion Matrix
- error: Class PreDiction Error
- class_report: Classification Report
- boundary: Decision Boundary
- rfe: Recursive Feature Selection
- learning: Learning Curve
- manifold: Manifold Learning
- calibration: Calibration Curve
- vc: Validation Curve
- dimension: Dimension Learning
- feature: Feature Importance
- feature_all: Feature Importance (All)
- parameter: Model Hyperparameter
- lift: Lift Curve
- gain: Gain Chart
- tree: Decision Tree
- ks: KS Statistic Plot
# 提取所有模型预测结果
models_results = s.pull()
models_results
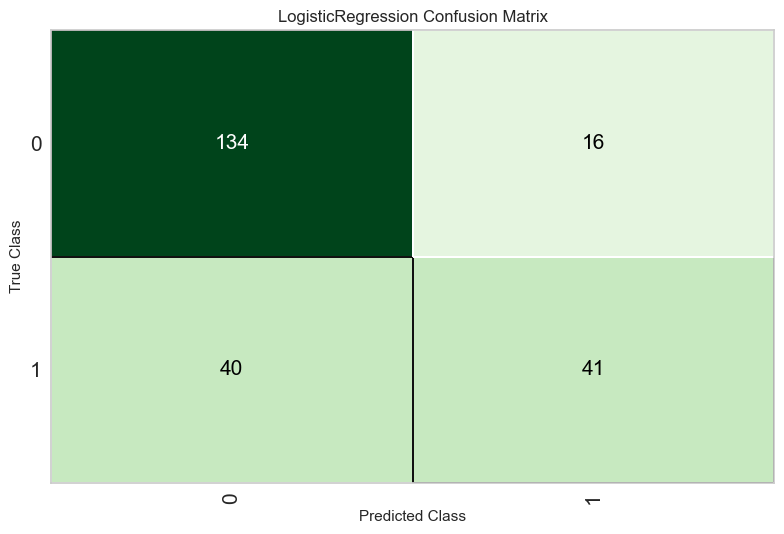
s.plot_model(best, plot = 'confusion_matrix')
如果在jupyter环境,可以通过evaluate_model函数来交互式展示模型的性能:
# s.evaluate_model(best)
模型预测
predict_model函数实现对数据进行预测,并返回包含预测标签prediction_label和分数prediction_score的Pandas表格。当data为None时,它预测测试集(在设置功能期间创建)上的标签和分数。
# 预测整个数据集
res = s.predict_model(best, data=data)
# 查看各行预测结果
# res
| Model | Accuracy | AUC | Recall | Prec. | F1 | Kappa | MCC | |
|---|---|---|---|---|---|---|---|---|
| 0 | Logistic Regression | 0.7708 | 0.8312 | 0.5149 | 0.7500 | 0.6106 | 0.4561 | 0.4723 |
# 预测用于数据训练的测试集
res = s.predict_model(best)
| Model | Accuracy | AUC | Recall | Prec. | F1 | Kappa | MCC | |
|---|---|---|---|---|---|---|---|---|
| 0 | Logistic Regression | 0.7576 | 0.8553 | 0.5062 | 0.7193 | 0.5942 | 0.4287 | 0.4422 |
模型保存与导入
# 保存模型到本地
_ = s.save_model(best, 'best_model', verbose = False)
# 导入模型
model = s.load_model( 'best_model')
# 查看模型结构
# model
Transformation Pipeline and Model Successfully Loaded
# 预测整个数据集
res = s.predict_model(model, data=data)
| Model | Accuracy | AUC | Recall | Prec. | F1 | Kappa | MCC | |
|---|---|---|---|---|---|---|---|---|
| 0 | Logistic Regression | 0.7708 | 0.8312 | 0.5149 | 0.7500 | 0.6106 | 0.4561 | 0.4723 |
1.2 回归
PyCaret提供了regression模型实现回归任务,regression模块与classification模块使用方法一致。
# 加载保险费用示例数据集
from pycaret.datasets import get_data
data = get_data(dataset='./datasets/insurance', verbose=False)
# 从网络下载
# data = get_data(dataset='insurance', verbose=False)
data.head()
# 创建数据管道
from pycaret.regression import RegressionExperiment
s = RegressionExperiment()
# 预测charges列
s.setup(data, target = 'charges', session_id = 0)
# 另一种数据管道创建方式
# from pycaret.regression import *
# s = setup(data, target = 'charges', session_id = 0)
| Description | Value | |
|---|---|---|
| 0 | Session id | 0 |
| 1 | Target | charges |
| 2 | Target type | Regression |
| 3 | Original data shape | (1338, 7) |
| 4 | Transformed data shape | (1338, 10) |
| 5 | Transformed train set shape | (936, 10) |
| 6 | Transformed test set shape | (402, 10) |
| 7 | Numeric features | 3 |
| 8 | Categorical features | 3 |
| 9 | Preprocess | True |
| 10 | Imputation type | simple |
| 11 | Numeric imputation | mean |
| 12 | Categorical imputation | mode |
| 13 | Maximum one-hot encoding | 25 |
| 14 | Encoding method | None |
| 15 | Fold Generator | KFold |
| 16 | Fold Number | 10 |
| 17 | CPU Jobs | -1 |
| 18 | Use GPU | False |
| 19 | Log Experiment | False |
| 20 | Experiment Name | reg-default-name |
| 21 | USI | eb9d |
<pycaret.regression.oop.RegressionExperiment at 0x200dedc2d30>
# 评估各类模型
best = s.compare_models()
| Model | MAE | MSE | RMSE | R2 | RMSLE | MAPE | TT (Sec) | |
|---|---|---|---|---|---|---|---|---|
| gbr | Gradient Boosting Regressor | 2723.2453 | 23787529.5872 | 4832.4785 | 0.8254 | 0.4427 | 0.3140 | 0.0550 |
| lightgbm | Light Gradient Boosting Machine | 2998.1311 | 25738691.2181 | 5012.2404 | 0.8106 | 0.5525 | 0.3709 | 0.1140 |
| rf | Random Forest Regressor | 2915.7018 | 26780127.0016 | 5109.5098 | 0.8031 | 0.4855 | 0.3520 | 0.0670 |
| et | Extra Trees Regressor | 2841.8257 | 28559316.9533 | 5243.5828 | 0.7931 | 0.4671 | 0.3218 | 0.0670 |
| ada | AdaBoost Regressor | 4180.2669 | 28289551.0048 | 5297.6817 | 0.7886 | 0.5935 | 0.6545 | 0.0210 |
| ridge | Ridge Regression | 4304.2640 | 38786967.4768 | 6188.6966 | 0.7152 | 0.5794 | 0.4283 | 0.0230 |
| lar | Least Angle Regression | 4293.9886 | 38781666.5991 | 6188.3301 | 0.7151 | 0.5893 | 0.4263 | 0.0210 |
| llar | Lasso Least Angle Regression | 4294.2135 | 38780221.0039 | 6188.1906 | 0.7151 | 0.5891 | 0.4264 | 0.0200 |
| br | Bayesian Ridge | 4299.8532 | 38785479.0984 | 6188.6026 | 0.7151 | 0.5784 | 0.4274 | 0.0200 |
| lasso | Lasso Regression | 4294.2186 | 38780210.5665 | 6188.1898 | 0.7151 | 0.5892 | 0.4264 | 0.0370 |
| lr | Linear Regression | 4293.9886 | 38781666.5991 | 6188.3301 | 0.7151 | 0.5893 | 0.4263 | 0.0350 |
| dt | Decision Tree Regressor | 3550.6534 | 51149204.9032 | 7095.9170 | 0.6127 | 0.5839 | 0.4537 | 0.0230 |
| huber | Huber Regressor | 3769.3076 | 53638697.2337 | 7254.7108 | 0.6095 | 0.4528 | 0.2187 | 0.0250 |
| par | Passive Aggressive Regressor | 4144.7180 | 62949698.1775 | 7862.7604 | 0.5433 | 0.4634 | 0.2465 | 0.0210 |
| en | Elastic Net | 7248.9376 | 89841235.9517 | 9405.5846 | 0.3534 | 0.7346 | 0.9238 | 0.0210 |
| omp | Orthogonal Matching Pursuit | 8916.1927 | 130904492.3067 | 11356.4120 | 0.0561 | 0.8781 | 1.1598 | 0.0180 |
| knn | K Neighbors Regressor | 8161.8875 | 137982796.8000 | 11676.3735 | -0.0011 | 0.8744 | 0.9742 | 0.0250 |
| dummy | Dummy Regressor | 8892.4478 | 141597492.8000 | 11823.4271 | -0.0221 | 0.9868 | 1.4909 | 0.0210 |
print(best)
GradientBoostingRegressor(alpha=0.9, ccp_alpha=0.0, criterion='friedman_mse',
init=None, learning_rate=0.1, loss='squared_error',
max_depth=3, max_features=None, max_leaf_nodes=None,
min_impurity_decrease=0.0, min_samples_leaf=1,
min_samples_split=2, min_weight_fraction_leaf=0.0,
n_estimators=100, n_iter_no_change=None,
random_state=0, subsample=1.0, tol=0.0001,
validation_fraction=0.1, verbose=0, warm_start=False)
1.3 聚类
ParCaret提供了clustering模块实现无监督聚类。
数据准备
# 导入珠宝数据集
from pycaret.datasets import get_data
# 根据数据集特征进行聚类
data = get_data('./datasets/jewellery')
# data = get_data('jewellery')
# 创建数据管道
from pycaret.clustering import ClusteringExperiment
s = ClusteringExperiment()
# normalize归一化数据
s.setup(data, normalize = True, verbose = False)
# 另一种数据管道创建方式
# from pycaret.clustering import *
# s = setup(data, normalize = True)
<pycaret.clustering.oop.ClusteringExperiment at 0x200dec86340>
模型创建
PyCaret在聚类任务中提供create_model选择合适的方法来构建聚类模型,而不是全部比较。
kmeans = s.create_model('kmeans')
| Silhouette | Calinski-Harabasz | Davies-Bouldin | Homogeneity | Rand Index | Completeness | |
|---|---|---|---|---|---|---|
| 0 | 0.7581 | 1611.2647 | 0.3743 | 0 | 0 | 0 |
create_model函数支持的聚类方法如下:
s.models()
print(kmeans)
# 查看聚类数
print(kmeans.n_clusters)
KMeans(algorithm='lloyd', copy_x=True, init='k-means++', max_iter=300,
n_clusters=4, n_init='auto', random_state=1459, tol=0.0001, verbose=0)
4
数据展示
# jupyter环境下交互可视化展示
# s.evaluate_model(kmeans)
# 结果可视化
# 'cluster' - Cluster PCA Plot (2d)
# 'tsne' - Cluster t-SNE (3d)
# 'elbow' - Elbow Plot
# 'silhouette' - Silhouette Plot
# 'distance' - Distance Plot
# 'distribution' - Distribution Plot
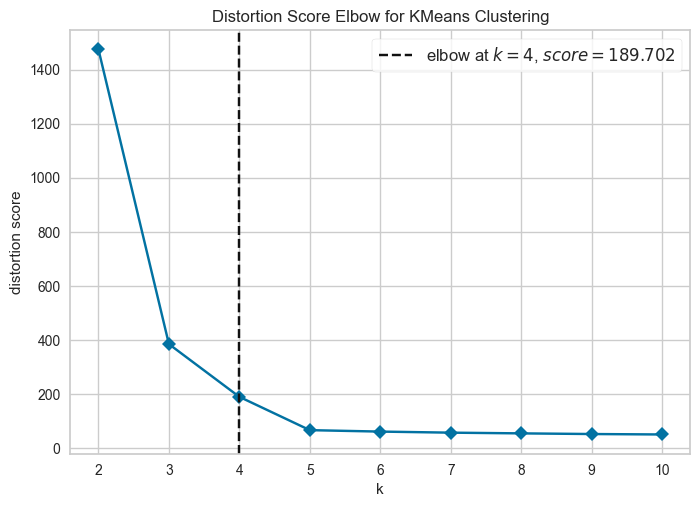
s.plot_model(kmeans, plot = 'elbow')
标签分配与数据预测
为训练数据分配聚类标签:
result = s.assign_model(kmeans)
result.head()
为新的数据进行标签分配:
predictions = s.predict_model(kmeans, data = data)
predictions.head()
1.4 异常检测
PyCaret的anomaly detection模块是一个无监督的机器学习模块,用于识别与大多数数据存在显著差异的罕见项目、事件或观测值。通常,这些异常项目会转化为某种问题,如**欺诈、结构缺陷、医疗问题或错误。anomaly detection模块的使用类似于cluster模块。
数据准备
from pycaret.datasets import get_data
data = get_data('./datasets/anomaly')
# data = get_data('anomaly')
from pycaret.anomaly import AnomalyExperiment
s = AnomalyExperiment()
s.setup(data, session_id = 0)
# 另一种加载方式
# from pycaret.anomaly import *
# s = setup(data, session_id = 0)
| Description | Value | |
|---|---|---|
| 0 | Session id | 0 |
| 1 | Original data shape | (1000, 10) |
| 2 | Transformed data shape | (1000, 10) |
| 3 | Numeric features | 10 |
| 4 | Preprocess | True |
| 5 | Imputation type | simple |
| 6 | Numeric imputation | mean |
| 7 | Categorical imputation | mode |
| 8 | CPU Jobs | -1 |
| 9 | Use GPU | False |
| 10 | Log Experiment | False |
| 11 | Experiment Name | anomaly-default-name |
| 12 | USI | 54db |
<pycaret.anomaly.oop.AnomalyExperiment at 0x200e14f5250>
模型创建
iforest = s.create_model('iforest')
print(iforest)
IForest(behaviour='new', bootstrap=False, contamination=0.05,
max_features=1.0, max_samples='auto', n_estimators=100, n_jobs=-1,
random_state=0, verbose=0)
anomaly detection模块所支持的模型列表如下:
s.models()
标签分配与数据预测
为训练数据分配聚类标签:
result = s.assign_model(iforest)
result.head()
为新的数据进行标签分配:
predictions = s.predict_model(iforest, data = data)
predictions.head()
1.5 时序预测
PyCaret时间序列预测Time Series模块支持多种预测方法,如ARIMA、Prophet和LSTM。它还提供了各种功能来处理缺失值、时间序列分解和数据可视化。
数据准备
# 乘客时序数据
from pycaret.datasets import get_data
# 下载路径:https://raw.GitHubusercontent.com/sktime/sktime/main/sktime/datasets/data/Airline/Airline.csv
data = get_data('./datasets/airline')
# data = get_data('airline')
import pandas as pd
data['Date'] = pd.to_datetime(data['Date'])
# 并将Date设置为列号
data.set_index('Date', inplace=True)
from pycaret.time_series import TSForecastingExperiment
s = TSForecastingExperiment()
# fh: 用于预测的预测范围。默认值设置为1,即预测前方一点。,fold: 交叉验证中折数
s.setup(data, fh = 3, fold = 5, session_id = 0, verbose = False)
# from pycaret.time_series import *
# s = setup(data, fh = 3, fold = 5, session_id = 0)
<pycaret.time_series.forecasting.oop.TSForecastingExperiment at 0x200dee26910>
模型训练与评估
best = s.compare_models()
| Model | MASE | RMSSE | MAE | RMSE | MAPE | SMAPE | R2 | TT (Sec) | |
|---|---|---|---|---|---|---|---|---|---|
| stlf | STLF | 0.4240 | 0.4429 | 12.8002 | 15.1933 | 0.0266 | 0.0268 | 0.4296 | 0.0300 |
| exp_smooth | ExPONEntial Smoothing | 0.5063 | 0.5378 | 15.2900 | 18.4455 | 0.0334 | 0.0335 | -0.0521 | 0.0500 |
| ets | ETS | 0.5520 | 0.5801 | 16.6164 | 19.8391 | 0.0354 | 0.0357 | -0.0740 | 0.0680 |
| arima | ARIMA | 0.6480 | 0.6501 | 19.5728 | 22.3027 | 0.0412 | 0.0420 | -0.0796 | 0.0420 |
| auto_arima | Auto ARIMA | 0.6526 | 0.6300 | 19.7405 | 21.6202 | 0.0414 | 0.0421 | -0.0560 | 10.5220 |
| theta | Theta Forecaster | 0.8458 | 0.8223 | 25.7024 | 28.3332 | 0.0524 | 0.0541 | -0.7710 | 0.0220 |
| huber_cds_dt | Huber w/ Cond. Deseasonalize & Detrending | 0.9002 | 0.8900 | 27.2568 | 30.5782 | 0.0550 | 0.0572 | -0.0309 | 0.0680 |
| knn_cds_dt | K Neighbors w/ Cond. Deseasonalize & Detrending | 0.9381 | 0.8830 | 28.5678 | 30.5007 | 0.0555 | 0.0575 | 0.0908 | 0.0920 |
| lr_cds_dt | Linear w/ Cond. Deseasonalize & Detrending | 0.9469 | 0.9297 | 28.6337 | 31.9163 | 0.0581 | 0.0605 | -0.1620 | 0.0820 |
| ridge_cds_dt | Ridge w/ Cond. Deseasonalize & Detrending | 0.9469 | 0.9297 | 28.6340 | 31.9164 | 0.0581 | 0.0605 | -0.1620 | 0.0680 |
| en_cds_dt | Elastic Net w/ Cond. Deseasonalize & Detrending | 0.9499 | 0.9320 | 28.7271 | 31.9952 | 0.0582 | 0.0606 | -0.1579 | 0.0700 |
| llar_cds_dt | Lasso Least Angular Regressor w/ Cond. Deseasonalize & Detrending | 0.9520 | 0.9336 | 28.7917 | 32.0528 | 0.0583 | 0.0607 | -0.1559 | 0.0560 |
| lasso_cds_dt | Lasso w/ Cond. Deseasonalize & Detrending | 0.9521 | 0.9337 | 28.7941 | 32.0557 | 0.0583 | 0.0607 | -0.1560 | 0.0720 |
| br_cds_dt | Bayesian Ridge w/ Cond. Deseasonalize & Detrending | 0.9551 | 0.9347 | 28.9018 | 32.1013 | 0.0582 | 0.0606 | -0.1377 | 0.0580 |
| et_cds_dt | Extra Trees w/ Cond. Deseasonalize & Detrending | 1.0322 | 0.9942 | 31.4048 | 34.3054 | 0.0607 | 0.0633 | -0.1660 | 0.1280 |
| rf_cds_dt | Random Forest w/ Cond. Deseasonalize & Detrending | 1.0851 | 1.0286 | 32.9791 | 35.4666 | 0.0641 | 0.0670 | -0.3545 | 0.1400 |
| lightgbm_cds_dt | Light Gradient Boosting w/ Cond. Deseasonalize & Detrending | 1.1409 | 1.1040 | 34.5999 | 37.9918 | 0.0670 | 0.0701 | -0.3994 | 0.0900 |
| ada_cds_dt | AdaBoost w/ Cond. Deseasonalize & Detrending | 1.1441 | 1.0843 | 34.7451 | 37.3681 | 0.0664 | 0.0697 | -0.3004 | 0.0920 |
| gbr_cds_dt | Gradient Boosting w/ Cond. Deseasonalize & Detrending | 1.1697 | 1.1094 | 35.4408 | 38.1373 | 0.0697 | 0.0729 | -0.4163 | 0.0900 |
| omp_cds_dt | Orthogonal Matching Pursuit w/ Cond. Deseasonalize & Detrending | 1.1793 | 1.1250 | 35.7348 | 38.6755 | 0.0706 | 0.0732 | -0.5095 | 0.0620 |
| dt_cds_dt | Decision Tree w/ Cond. Deseasonalize & Detrending | 1.2704 | 1.2371 | 38.4976 | 42.4846 | 0.0773 | 0.0814 | -1.0382 | 0.0860 |
| snaive | Seasonal Naive Forecaster | 1.7700 | 1.5999 | 53.5333 | 54.9143 | 0.1136 | 0.1211 | -4.1630 | 0.1580 |
| naive | Naive Forecaster | 1.8145 | 1.7444 | 54.8667 | 59.8160 | 0.1135 | 0.1151 | -3.7710 | 0.1460 |
| polytrend | Polynomial Trend Forecaster | 2.3154 | 2.2507 | 70.1138 | 77.3400 | 0.1363 | 0.1468 | -4.6202 | 0.1080 |
| croston | Croston | 2.6211 | 2.4985 | 79.3645 | 85.8439 | 0.1515 | 0.1684 | -5.2294 | 0.0140 |
| grand_means | Grand Means Forecaster | 7.1261 | 6.3506 | 216.0214 | 218.4259 | 0.4377 | 0.5682 | -59.2684 | 0.1400 |
数据展示
# jupyter环境下交互可视化展示
# plot参数支持:
# - 'ts' - Time Series Plot
# - 'train_test_split' - Train Test Split
# - 'cv' - Cross Validation
# - 'acf' - Auto Correlation (ACF)
# - 'pacf' - Partial Auto Correlation (PACF)
# - 'decomp' - Classical Decomposition
# - 'decomp_stl' - STL Decomposition
# - 'diagnostics' - Diagnostics Plot
# - 'diff' - Difference Plot
# - 'periodogram' - Frequency Components (Periodogram)
# - 'fft' - Frequency Components (FFT)
# - 'ccf' - Cross Correlation (CCF)
# - 'forecast' - "Out-of-Sample" Forecast Plot
# - 'insample' - "In-Sample" Forecast Plot
# - 'residuals' - Residuals Plot
# s.plot_model(best, plot = 'forecast', data_kwargs = {'fh' : 24})
数据预测
# 使模型拟合包括测试样本在内的完整数据集
final_best = s.finalize_model(best)
s.predict_model(best, fh = 24)
s.save_model(final_best, 'final_best_model')
Transformation Pipeline and Model Successfully Saved
(ForecastingPipeline(steps=[('forecaster',
TransformedTargetForecaster(steps=[('model',
ForecastingPipeline(steps=[('forecaster',
TransformedTargetForecaster(steps=[('model',
STLForecaster(sp=12))]))]))]))]),
'final_best_model.pkl')
2 数据处理与清洗
2.1 缺失值处理
各种数据集可能由于多种原因存在缺失值或空记录。移除具有缺失值的样本是一种常见策略,但这会导致丢失可能有价值的数据。一种可替代的策略是对缺失值进行插值填充。在setup函数中可以指定如下参数,实现缺失值处理:
- imputation_type:取值可以是 'simple' 或 'iterative'或 None。当imputation_type设置为 'simple' 时,PyCaret 将使用简单的方式(numeric_imputation和categorical_imputation)对缺失值进行填充。而当设置为 'iterative' 时,则会使用模型估计的方式(numeric_iterative_imputer,categorical_iterative_imputer)进行填充处理。如果设置为 None,则不会执行任何缺失值填充操作
-
numeric_imputation: 设置数值类型的缺失值,方式如下:
- mean: 用列的平均值填充,默认
- drop: 删除包含缺失值的行
- median: 用列的中值填充
- mode: 用列最常见值填充
- knn: 使用K-最近邻方法拟合
- int or float: 用指定值替代
-
categorical_imputation:
- mode: 用列最常见值填充,默认
- drop: 删除包含缺失值的行
- str: 用指定字符替代
- numeric_iterative_imputer: 使用估计模型拟合值,可输入str或sklearn模型, 默认使用lightgbm
- categorical_iterative_imputer: 使用估计模型差值,可输入str或sklearn模型, 默认使用lightgbm
加载数据
# load dataset
from pycaret.datasets import get_data
# 从本地加载数据,注意dataset是数据的文件名
data = get_data(dataset='./datasets/hepatitis', verbose=False)
# data = get_data('hepatitis',verbose=False)
# 可以看到第三行STEROID列出现NaN值
data.head()
# 使用均值填充数据
from pycaret.classification import ClassificationExperiment
s = ClassificationExperiment()
# 均值
# s.data['STEROID'].mean()
s.setup(data = data, session_id=0, target = 'Class',verbose=False,
# 设置data_split_shuffle和data_split_stratify为False不打乱数据
data_split_shuffle = False, data_split_stratify = False,
imputation_type='simple', numeric_iterative_imputer='drop')
# 查看转换后的数据
s.get_config('dataset_transformed').head()
# 使用knn拟合数据
from pycaret.classification import ClassificationExperiment
s = ClassificationExperiment()
s.setup(data = data, session_id=0, target = 'Class',verbose=False,
# 设置data_split_shuffle和data_split_stratify为False不打乱数据
data_split_shuffle = False, data_split_stratify = False,
imputation_type='simple', numeric_imputation = 'knn')
# 查看转换后的数据
s.get_config('dataset_transformed').head()
# 使用lightgbmn拟合数据
# from pycaret.classification import ClassificationExperiment
# s = ClassificationExperiment()
# s.setup(data = data, session_id=0, target = 'Class',verbose=False,
# # 设置data_split_shuffle和data_split_stratify为False不打乱数据
# data_split_shuffle = False, data_split_stratify = False,
# imputation_type='iterative', numeric_iterative_imputer = 'lightgbm')
# 查看转换后的数据
# s.get_config('dataset_transformed').head()
2.2 类型转换
虽然 PyCaret具有自动识别特征类型的功能,但PyCaret提供了数据类型自定义参数,用户可以对数据集进行更精细的控制和指导,以确保模型训练和特征工程的效果更加符合用户的预期和需求。这些自定义参数如下:
- numeric_features:用于指定数据集中的数值特征列的参数。这些特征将被视为连续型变量进行处理
- categorical_features:用于指定数据集中的分类特征列的参数。这些特征将被视为离散型变量进行处理
- date_features:用于指定数据集中的日期特征列的参数。这些特征将被视为日期型变量进行处理
- create_date_columns:用于指定是否从日期特征中创建新的日期相关列的参数
- text_features:用于指定数据集中的文本特征列的参数。这些特征将被视为文本型变量进行处理
- text_features_method:用于指定对文本特征进行处理的方法的参数
- ignore_features:用于指定在建模过程中需要忽略的特征列的参数
- keep_features:用于指定在建模过程中需要保留的特征列的参数
# 转换变量类型
from pycaret.datasets import get_data
data = get_data(dataset='./datasets/hepatitis', verbose=False)
from pycaret.classification import *
s = setup(data = data, target = 'Class', ignore_features = ['SEX','AGE'], categorical_features=['STEROID'],verbose = False,
data_split_shuffle = False, data_split_stratify = False)
# 查看转换后的数据,前两列消失,STEROID变为分类变量
s.get_config('dataset_transformed').head()
2.3 独热编码
当数据集中包含分类变量时,这些变量通常需要转换为模型可以理解的数值形式。独热编码是一种常用的方法,它将每个分类变量转换为一组二进制变量,其中每个变量对应一个可能的分类值,并且只有一个变量在任何给定时间点上取值为 1,其余变量均为 0。可以通过传递参数categorical_features来指定要进行独热编码的列。例如:
# load dataset
from pycaret.datasets import get_data
data = get_data(dataset='./datasets/pokemon', verbose=False)
# data = get_data('pokemon')
data.head()
# 对Type 1实现独热编码
len(set(data['Type 1']))
18
from pycaret.classification import *
s = setup(data = data, categorical_features =["Type 1"],target = 'Legendary', verbose=False)
# 查看转换后的数据Type 1变为独热编码
s.get_config('dataset_transformed').head()
2.4 数据平衡
在 PyCaret 中,fix_imbalance 和 fix_imbalance_method 是用于处理不平衡数据集的两个参数。这些参数通常用于在训练模型之前对数据集进行预处理,以解决类别不平衡问题。
- fix_imbalance 参数:这是一个布尔值参数,用于指示是否对不平衡数据集进行处理。当设置为 True 时,PyCaret 将自动检测数据集中的类别不平衡问题,并尝试通过采样方法来解决。当设置为 False 时,PyCaret 将使用原始的不平衡数据集进行模型训练
-
fix_imbalance_method 参数:这是一个字符串参数,用于指定处理不平衡数据集的方法。可选的值包括:
- 使用 SMOTE(Synthetic Minority Over-sampling Technique)来生成人工合成样本,从而平衡类别(默认参数smote)
- 使用 imbalanced-learn 提供的估算模型
# 加载数据
from pycaret.datasets import get_data
data = get_data(dataset='./datasets/credit', verbose=False)
# data = get_data('credit')
data.head()
# 查看数据各类别数
category_counts = data['default'].value_counts()
category_counts
default
0 18694
1 5306
Name: count, dtype: int64
from pycaret.classification import *
s = setup(data = data, target = 'default', fix_imbalance = True, verbose = False)
# 可以看到类1数据量变多了
s.get_config('dataset_transformed')['default'].value_counts()
default
0 18694
1 14678
Name: count, dtype: int64
2.5 异常值处理
PyCaret的remove_outliers函数可以在训练模型之前识别和删除数据集中的异常值。它使用奇异值分解技术进行PCA线性降维来识别异常值,并可以通过setup中的outliers_threshold参数控制异常值的比例(默认0.05)。
from pycaret.datasets import get_data
data = get_data(dataset='./datasets/insurance', verbose=False)
# insurance = get_data('insurance')
# 数据维度
data.shape
(1338, 7)
from pycaret.regression import *
s = setup(data = data, target = 'charges', remove_outliers = True ,verbose = False, outliers_threshold = 0.02)
# 移除异常数据后,数据量变少
s.get_config('dataset_transformed').shape
(1319, 10)
2.6 特征重要性
特征重要性是一种用于选择数据集中对预测目标变量最有贡献的特征的过程。与使用所有特征相比,仅使用选定的特征可以减少过拟合的风险,提高准确性,并缩短训练时间。在PyCaret中,可以通过使用feature_selection参数来实现这一目的。对于PyCaret中几个与特征选择相关参数的解释如下:
- feature_selection:用于指定是否在模型训练过程中进行特征选择。可以设置为 True 或 False。
-
feature_selection_method:特征选择方法:
- 'univariate': 使用sklearn的SelectKBest,基于统计测试来选择与目标变量最相关的特征。
- 'classic(默认)': 使用sklearn的SelectFromModel,利用监督学习模型的特征重要性或系数来选择最重要的特征。
- 'sequential': 使用sklearn的SequentialFeatureSelector,该类根据指定的算法(如前向选择、后向选择等)以及性能指标(如交叉验证得分)逐步选择特征。
- n_features_to_select:特征选择的最大特征数量或比例。如果<1,则为起始特征的比例。默认为0.2。该参数在计数时不考虑 ignore_features 和 keep_features 中的特征。
from pycaret.datasets import get_data
data = get_data('./datasets/diabetes')
from pycaret.regression import *
# feature_selection选择特征, n_features_to_select选择特征比例
s = setup(data = data, target = 'Class variable', feature_selection = True, feature_selection_method = 'univariate',
n_features_to_select = 0.3, verbose = False)
# 查看哪些特征保留下来
s.get_config('X_transformed').columns
s.get_config('X_transformed').head()
2.7 归一化
数据归一化
在 PyCaret 中,normalize 和 normalize_method 参数用于数据预处理中的特征缩放操作。特征缩放是指将数据的特征值按比例缩放,使之落入一个小的特定范围,这样可以消除特征之间的量纲影响,使模型训练更加稳定和准确。下面是关于这两个参数的说明:
- normalize: 这是一个布尔值参数,用于指定是否对特征进行缩放。默认情况下,它的取值为 False,表示不进行特征缩放。如果将其设置为 True,则会启用特征缩放功能。
-
normalize_method: 这是一个字符串参数,用于指定特征缩放的方法。可选的值有:
- zscore(默认): 使用 Z 分数标准化方法,也称为标准化或 Z 标准化。该方法将特征的值转换为其 Z 分数,即将特征值减去其均值,然后除以其标准差,从而使得特征的均值为 0,标准差为 1。
- minmax: 使用 Min-Max 标准化方法,也称为归一化。该方法将特征的值线性转换到指定的最小值和最大值之间,默认情况下是 [0, 1] 范围。
- maxabs: 使用 MaxAbs 标准化方法。该方法将特征的值除以特征的最大绝对值,将特征的值缩放到 [-1, 1] 范围内。
- robust: 使用 RobustScaler 标准化方法。该方法对数据的每个特征进行中心化和缩放,使用特征的中位数和四分位数范围来缩放特征。
from pycaret.datasets import get_data
data = get_data('./datasets/pokemon')
data.head()
# 归一化
from pycaret.classification import *
s = setup(data, target='Legendary', normalize=True, normalize_method='robust', verbose=False)
数据归一化结果:
s.get_config('X_transformed').head()
特征变换
归一化会重新调整数据,使其在新的范围内,以减少方差中幅度的影响。特征变换是一种更彻底的技术。通过转换改变数据的分布形状,使得转换后的数据可以被表示为正态分布或近似正态分布。PyCaret中通过transformation参数开启特征转换,transformation_method设置转换方法:yeo-johnson(默认)和分位数。此外除了特征变换,还有目标变换。目标变换它将改变目标变量而不是特征的分布形状。此功能仅在pycarte.regression模块中可用。使用transform_target开启目标变换,transformation_method设置转换方法。
from pycaret.classification import *
s = setup(data = data, target = 'Legendary', transformation = True, verbose = False)
# 特征变换结果
s.get_config('X_transformed').head()
3 参考
- pycaret
- pycaret-docs
- pycaret-datasets
- lightgbm
- cuml
- imbalanced-learn











